O ácido desoxirribonucleico (ADN, em português: ácido desoxirribonucleico; ou DNA, em inglês: deoxyribonucleic acid), é um composto orgânico cujas moléculas contêm as instruções genéticas que coordenam o desenvolvimento e funcionamento de todos os seres vivos e alguns vírus. O seu principal papel é armazenar as informações necessárias para a construção das proteínas e ARNs. Os segmentos de ADN que são responsáveis por carregar a informação genética são denominados genes. O restante da seqüência de ADN tem importância estrutural ou está envolvido na regulação do uso da informação genética.
A estrutura da molécula de ADN foi descoberta conjuntamente pelo estadunidense James Watson e pelo britânico Francis Crick em 7 de Março de 1953, o que lhes valeu o Prêmio Nobel de Fisiologia ou Medicina em 1962, juntamente com Maurice Wilkins.
Do ponto de vista químico, o ADN é um longo polímero de unidades simples (monômeros) de nucleotídeos, cujo cerne é formado por moléculas de açúcares e fosfato intercalados unidos por ligações fosfodiéster. Ligada à molécula de açúcar está uma de quatro bases nitrogenadas e é a seqüência dessas bases ao longo da molécula de ADN que carrega a informação genética. A leitura destas seqüências é feita através do código genético, o qual especifica a sequência linear dos aminoácidos das proteínas. A tradução é feita por um RNA mensageiro que copia parte da cadeia de ADN por um processo chamado transcrição e posteriormente a informação contida neste é "traduzida" em proteínas pela tradução. Embora a maioria do ARN produzido seja usado na síntese de proteínas, algum ARN tem função estrutural, como por exemplo o ARN ribossômico, que faz parte da constituição dos ribossomos.
Dentro da célula, o ADN pode ser observado numa estrutura chamada cromossoma durante a metafase e o conjunto de cromossomas de uma célula forma o cariótipo. Antes da divisão celular os cromossomas são duplicados através de um processo chamado replicação. Eucariontes como animais, plantas e fungos têm o seu ADN dentro do núcleo enquanto que procariontes como as bactérias o têm disperso no citoplasma. Dentro dos cromossomas, proteínas da cromatina como as histonas compactam e organizam o ADN. Estas estruturas compactas guiam as interações entre o ADN e outras proteínas, ajudando a controlar que partes do ADN são transcritas.
O ADN é responsável pela transmissão das características hereditárias de cada espécie de ser vivo.
História
Até 1899
|
1865 - Gregor Mendel publica trabalho sobre experimentos com ervilhas em que propõe as leis da hereditariedade e supõe que as características hereditárias são transmitidas em unidades. |
|
1869 - O suíço Friedrich Miescher isola, a partir do pus humano e do esperma do salmão, uma substância com alto teor de fósforo que chama de "nucleína", posteriormente denominada "ácido desoxirribonucléico" (ADN). |
|
1882 - O alemão Walter Flemming descobre corpos com formato de bastão dentro do núcleo das células, que denomina "cromossomas". |
1900 até 1919
|
1900 - O holandês Hugo de Vries, o alemão Carl Correns e o austríaco Erich Tschermak von Seysenegg chegam de forma independente aos resultados de Mendel sobre as leis da hereditariedade. |
|
1902 - O norte-americano Walter Sutton e o alemão Theodor Boveri dão início à teoria cromossómica da hereditariedade. |
|
1909 - O dinamarquês Wilhelm Johannsen introduz o termo "gene" para descrever a unidade mendeliana da hereditariedade. Utiliza os termos "genótipo" e "fenótipo" para diferenciar as características genéticas de um indivíduo de sua aparência externa. |
|
1915 - O norte-americano Thomas Hunt Morgan e outros publicam o livro "O Mecanismo da Hereditariedade Mendeliana", no qual relatam experimentos com drosófilas e mostram que os genes estão linearmente dispostos nos cromossomos. |
1940 até 1959
|
1949 - O austríaco Erwin Chargaff descobre, nos EUA, uma relação quantitativa entre as bases do ADN: a proporção entre adenina e timina é sempre igual, e o mesmo ocorre entre guanina e citosina. |
|
1950 - Os norte-americanos Linus Pauling e Robert Corey identificam a estrutura molecular básica de proteínas. Eles propõem uma estrutura para o ADN com três cadeias helicoidais entrelaçadas (o modelo da tripla hélice). |
|
1952 - A britânica Rosalind Franklin obtém imagens de ADN , por difração de raios X. |
|
1953 - O norte-americano James Watson e o britânico Francis Crick decifram, em 7 de Março, a estrutura de dupla hélice para o ADN e a publicam na revista "Nature" de 25 de Abril. Em 30 de maio, também na "Nature", Watson e Crick analisam as implicações genéticas de seu modelo e sugerem um mecanismo para a replicação do ADN. |
James Watson
|
1958 - Os norte-americanos Matthew Meselson e Franklin Stahl confirmam a hipótese feita por Watson e Crick de que o ADN replica-se de maneira semiconservativa. |
1960 até 1979
|
1972 - O norte-americano Paul Berg obtém moléculas de ADN recombinante, unindo ADN de diferentes espécies e inserindo esse ADN híbrido em uma célula hospedeira. |
|
1975 - Grupos de pesquisa desenvolvem métodos de seqüenciamento de ADN. |
|
1976 - Criada a primeira companhia de engenharia genética, a Genentech. |
1980 até 1999
|
1980 - A Suprema Corte dos EUA decide que formas de vida alteradas podem ser patenteadas. |
|
1982 - O primeiro animal (camundongo) transgênico é obtido nos EUA. |
|
1983 - Companhias nos EUA conseguem obter patentes para plantas geneticamente modificadas. É mapeado nos EUA o primeiro gene relacionado a uma doença, um marcador da doença de Huntington encontrado no cromossoma 4. |
|
1985 - Alec Jeffreys (inglês) descreve técnica de identificação que ficou conhecida como "impressão digital" por ADN. |
Os NIH dos EUA aprovam diretrizes gerais para a realização de experimentos com terapia genética em seres humanos.
|
1986 - Plantas de tabaco geneticamente modificadas para se tornarem resistentes a herbicida são testadas em campo pela primeira vez, nos EUA e na França. |
Ovelha Dolly, empalhada no Royal Museum of Scothand, em Edingurgo
|
1988 - Nos EUA, Philip Leder e Timothy Stewart obtêm primeira patente para um animal geneticamente modificado, um camundongo. |
|
1989 - Criação nos EUA do Instituto Nacional para Pesquisa do Genoma Humano (NHGRI), chefiado por James Watson, para determinar toda a sequência do ADN que compõe os cromossomas humanos. |
|
1994 - Liberação de tomate, primeiro alimento geneticamente modificado cuja venda é aprovada pela FDA. |
|
1995 - É obtida a primeira sequência completa de ADN de um organismo de vida livre, a bactéria Hemophilus influenzae. |
|
1997 - Nascimento da ovelha Dolly, primeiro mamífero clonado a partir de uma célula de um animal adulto pelo Instituto Roslin (Escócia). Mapa genético completo do camundongo. |
2000 até 2019
|
2000 - Pesquisadores do consórcio público Projeto Genoma Humano e da empresa privada norte-americana Celera anunciam o rascunho do genoma humano. No Brasil, pesquisadores paulistas anunciam o seqüenciamento do genoma da bactéria Xylella fastidiosa, a causadora da doença do amarelinho em cítricos. O artigo foi destacado na capa da revista "Nature". |
|
12 de Fevereiro de 2001 - É anunciada a publicação da análise da sequência do genoma humano. |
|
2003 - Conclusão parcial da análise da sequência do genoma humano (PGH). |
Descoberta
Friedrich Miescher
A história do DNA começa no final da década de 1860, com a chegada do médico suiço Friedrich Miescher (1844-1895) à Universidade de Tübingen, uma pacata cidade no sul da Alemanha. O jovem pesquisador estava disposto à dedicar-se ao estudo da química da célula e escolheu essa universidade porque nela o químico Felix Hoppe-Seyler (1825-1895) havia inaugurado um importante laboratório de química fisiológica. Na época floresciam idéias a respeito das origens e das funções das células. Há pouco tempo, a teoria da geração espontânea havia sido definitivamente desacreditada. A teoria celular estabelecia-se como um dos pilares da Biologia. Por tudo isso, as células atraíam a atenção de estudantes entusiasmados, como Miescher.
Felix Hoppe-Seyler foi quem primeiro descreveu as interações entre a hemoglobina, a proteína responsável pela cor do sangue, e o gás oxigênio. Seu trabalho levou-o a interessar-se aos glóbulos brancos presentes na circulação sangüínea. Foi por sugestão de Hoppe-Seyler que Miescher começou a estudar a química das células do pus; o material para a pesquisa era abundante, pois dezenas de bandagens com material purulento eram diariamente descartadas por um hospital próximo à universidade. Miescher trabalhou para desenvolver técnicas adequadas à retiração das células de pus das bandagens e à preparação para a análise química. O objetivo inicial era investigar as proteínas celulares, um grupo de substâncias descoberto cerca de 30 anos antes.
Em um dos seus muitos experimentos com células do pus, Miescher obteve um precipitado que diferia quimicamente de todas as substâncias protéicas conhecidas. Ele descobriu que a nova substância se concentrava no núcleo celular, na época considerado uma estrutura de pouca importância para o funcionamento celular. Aprimorando os métodos de extração e purificação da nova substância, Miescher demostrou que, além de estar nas células do pus, ela também estava presente em materiais tão diversos quanto o rim, o fígado , o testículo, a levedura e as hemácias nucleadas das aves.
A análise química mostrou que as quantidades relativas dos elementos hidrogênio (H), carbono (C), oxigênio (O) e nitrogênio (N) presentes diferiam das encontradas em proteínas; além disso, à substância descoberta Miescher denominou-a nucleína, pelo fato de ela estar concentrada no núcleo das células.
O trabalho sobre nucleína só foi publicado em 1871, após certa resistência do editor da revista científica, o próprio Hoppe-Seyler, que, no início, não acreditou nos resultados apresentados por Miescher. Mesmo depois da publicação do trabalho, muitos pesquisadores continuaram duvidando da existência da nucleína; na opinião deles, o achado de Miescher devia ser uma mistura de fosfato inorgânico e proteínas.[1][2]
Elucidação da composição química
As desconfianças quanto à real existência da nova substância descrita por Miescher só foram superadas por volta de 1889, quando Richard Altmann (1852-1900) obteve preparações altamente purificadas de nucleína, sem nenhuma contaminação por proteínas. Pelo fato de a substância ter caráter ácido, o que já havia sido detectado por Miescher, Altmann sugeriu que ela fosse chamada de ácido nucléico em vez de nucleína.
Outro pesquisador pioneiro na descoberta foi Albrecht Kossel (1853-1927). Em 1877, ele juntou-se ao grupo de pesquisa de Hoppe-Seyler, então trabalhando na Universidade de Estrasburgo (França), e começou a estudar a composição química das nucleínas. Kossel detectou dois tipos de bases nitrogenadas já conhecidas, a adenina e a guanina. Em 1893, identificou uma nova base nitrogenada, que era liberada pela degradação de nucleína da células do timo; por isso denominou-a timina. Logo em seguida, descobriu que a nucleína continha um quarto tipo de base nitrogenada, a qual denominou citosina. Em 1894, o grupo liderado por Kossel descobriu que os ácidos nucleicos continham também pentose, um açúcar com cinco átomos de carbono.
Em 1909, Phoebis Levine e Walter Jacobs (1883-1967) conseguiram determinar a organização das moléculas de fosfato, de pentose e base nitrogenada no ácido nucléico. Esses três componentes estão unidos entre si formando uma unidade fundamental, o nucleotídeo. Em 1930, Levine e colaboradores identificaram pentoses componente do ácido nucléico das células do timo, que denominaram 2-deoxi-D-ribose, pelo fato de ela possuir, no carbono 2 de sua cadeia, um átomo de oxigênio a menos que a ribose, uma pentose já conhecida, encontrada pelos pesquisadores em dois tipos de ácidos nucléicos: o ácido ribonucléico, ou ribose, e o ácido desoxirribonucléico, ou DNA, cujo açúcar é a desoxirribose.
Descoberta da transformação
Frederick Griffith em 1936
Frederick Griffith fez uma importante observação no curso dos experimentos com a bactéria Streptococcus pneumoniae em 1928. Esta bactéria, que causa a pneumonia nos humanos, normalmente é letal em camundongos. Entretanto algumas linhagens desta espécie de bactérias desenvolviam-se menos virulentas (menos capazes de causar doenças ou morte). Nos experimentos de Griffith, ele usou duas linhagens que são distinguíveis pelo surgimento de suas colônias quando cultivadas em laboratório. Uma linhagem era um tipo normalmente virulento e mortal para a maioria dos animais de laboratório. As células desta linhagem estão envoltas em uma cápsula de polissacarídeo, dando às colônias em aspecto liso, donde esta linhagem ser identificada com S. A outra linhagem de Griffith era um tipo mutante não virulento que crescia em camundongos mas sem ser letal. Nesta linhagem, a capa de polissacarídeo está ausente, dando às colônias um aspecto rugoso . Esta linhagem é chamada R.
Griffith matou algumas células virulentas, fervendo-as. Ele então injetou as células mortas por aquecimento nos camundongos. Os camundongos sobreviveram, mostrando que os restos das células não causam morte. Entretanto os camundongos injetados com uma mistura de células não virulenta mortas por aquecimento e células não virulentas vivas, morreram. Além disso, as células vivas podiam ser recuperadas de camundongos mortos. Estas células deram colônias lisas e foram virulentas em uma injeção subseqüente. De algum modo, os restos das células S aquecidas haviam convertido células R vivas em células S vivas.[3]
Streptococcus pneumoniae
A etapa seguinte era determinar que componente químico das células doadoras mortas havia causado esta transformação. Esta substância tinha mudado o genótipo da linhagem receptora e portanto podia ser uma candidata ao material genético. Este problema foi resolvido pelos experimentos feitos em 1944 por Oswald Avery e dois colegas, C M. Macleod e M. McCarty. Seu enfoque ao problema foi destruir quimicamente todas as principais categorias de substâncias no extrato das células mortas, uma de cada vez, e descobrir se o extrato havia perdido a habilidade em transformar. As células virulentas tinham uma capa lisa de polissacarídeo, enquanto as células não virulentas, não. Assim os polissacarídeos eram um candidato óbvio a ser o agente transformante. Entretanto, quando os polissacarídeos foram destruídos, a mistura ainda podia transformar. As proteínas, gorduras e ácido ribonucleico (RNA) foram todos demostrados como não sendo o agente transformante. A mistura só perdia a sua habilidade transformante quando a mistura doadora era tratada com enzima desoxirribonuclease (DNase), que quebra o DNA. Estes resultados indicavam fortemente que o DNA era o material genético. Hoje sabemos que os fragmentos do DNA transformante que conferem virulência entram no cromossomo bacteriano e substituem suas contrapartes que confere não virulência.[4]
Experimento de Hershey-Chase
Alfred D. Hershey em 1969
Os experimentos feitos por Avery e seus colegas foram definitivos, mas muitos cientistas mostraram-se muito relutantes em aceitar o DNA (e não as proteínas) como material genético. Evidências adicionais foram dadas em 1952 por Alfred Day Hershey e Martha Chase. O experimento de ambos usou o fago T2, um vírus que infecta na bactéria a informação específica que dita a reprodução de novas partículas virais. Se eles pudessem descobrir que material o fago estava injetando na bactéria hospedeira, determinariam o material genético do fago.
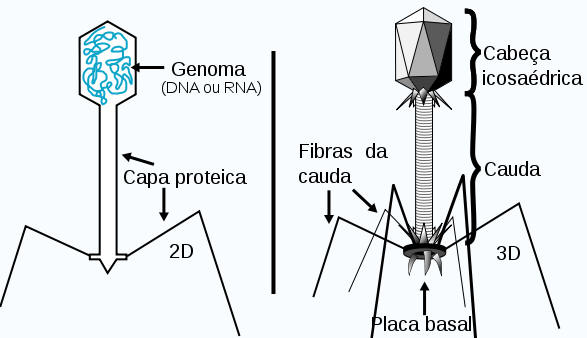
O fago tem uma constituição molecular relativamente simples. A maior parte de sua estrutura é de proteína, com o DNA contido dentro da capa de proteína de sua "cabeça". Hershey e Chase decidiram marcar o DNA e a proteína usando radioisótopos, de modo que pudessem rastrear os dois materiais durante a infecção. O fósforo não é encontrado nas proteínas mas é uma parte integrante do DNA. Contrariamente, o enxofre está presente nas proteínas mas nunca no DNA. Hershey e Chase incorporaram o radioisótopo de fósforo (32P) no DNA do fago e o enxofre (35S) nas proteínas de uma cultura separada de fagos. Eles então infectaram duas culturas de E. cume com muitas partículas de vírus por células: uma cultura de E. coli recebeu fagos marcados com 32P e a outra recebeu fagos marcados com 35S. Após dar tempo suficiente para que ocorresse a infecção, os cientistas removeram as embalagens de fago (chamadas ghosts) das células bacterianas por agitação em um liquidificador. Eles separaram as células bacterianas dos envoltórios dos fagos em uma centrífuga e então dosaram a radioatividade nas duas frações. Quando o fago marcado com 32P foi usado para infectar E. coli, a maioria da radioatividade foi encontrada dentro das bactérias, indicando que o DNA do fago havia entrado nas células. Quando era usado o fago marcado com 35S, maior parte do material radioativo estava nas capas dos fagos, indicando que a proteína do fago nunca entrava nas bactérias. A conclusão era inevitável: o DNA era o material hereditário. As proteínas do fago eram apenas embalagens estruturais abandonadas após o DNA viral entrar na bactéria.
Propriedades físicas e químicas
O ADN é um longo polímero formado por unidades repetidas chamadas nucleotídeos. [5] [6] A cadeia de ADN tem 2,2 a 2,4 nanômetros de largura, e um nucleotídeo possui aproximadamente 0,33 nanômetros de comprimento [7]. Embora os monômeros (nucleotídeos) que constituem o ADN sejam muito pequenos, polímeros de ADN podem ser moléculas enormes, com milhões de nucleotídeos. Por exemplo, o maior cromossomo humano (cromossomo 1), possui 220 milhões de pares de bases de comprimento. [8]
Em organismos vivos, o ADN não existe como uma molécula única (cadeia simples), mas sim como um par de moléculas firmemente associadas. [9] [10] As duas longas cadeias de ADN enrolam-se como uma trepadeira formando uma dupla hélice. Os nucleotídeos estão presentes em ambas as cadeias da dupla hélice, unidos com nucleotídeos da mesma cadeia por ligações fosfodiéster e à cadeia complementar através de pontes de hidrogênio formadas pelas suas bases. Em geral, uma base ligada a um açúcar é chamada nucleotídeo e uma base ligada a um açúcar e um ou mais fosfatos é chamada nucleotídeo. Portanto, o ADN pode ser referido como um polinucleotídeo. [11]
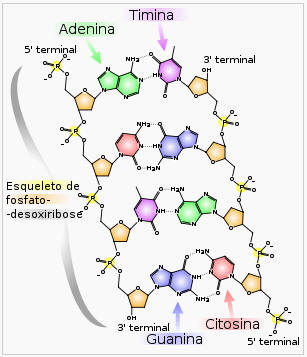
O cerne (backbone) da cadeia de ADN é formado por fosfato e resíduos de açúcar, dispostos alternadamente. O açúcar no ADN é 2-desoxirribose, e é uma pentose (açúcar com cinco carbonos). Os açúcares são unidos por grupos de fosfato que formam ligações fosfodiester entre o terceiro e quinto átomos de carbono dos anéis de açúcar adjacentes. Estas ligações assimétricas significam que uma cadeia de ADN tem uma direção. Numa dupla hélice, a direção dos nucleotídeos de uma cadeia é oposta à direção dos nucleotídeos da outra cadeia. O formato das cadeia do ADN é designado antiparalelo. As terminações assimétricas das cadeias de ADN são designadas terminais 5’ (cinco linha) e 3’ (três linha). Uma das diferenças principais entre o ADN e o ARN encontra-se no açúcar, com a substituição da 2-desoxirribose no ADN pela ribose no ARN.
A dupla hélice do ADN é estabilizada por pontes de hidrogênio entre as bases presas às duas cadeias. As quatro bases encontradas no ADN são a adenina (A), citosina (C), guanina (G) e timina (T). Estas quatro bases estão representadas na figura ao lado e ligam-se ao açúcar/fosfato para formar o nucleotídeo completo, que na figura é mostrado como adenosina monofosfato.
Estas bases são classificadas em dois tipos; a adenina e guanina são compostos heterocíclicos chamados purinas, enquanto que a citosina e timina são pirimidinas. Uma quinta base (uma pirimidina) chamada uracila (U) aparece no ARN e substitui a timina, a uracila difere da timina pela falta de um grupo de metila no seu anel. A uracila normalmente não está presente no ADN, só ocorrendo como um produto da decomposição da citosina. Uma raríssima exceção para esta regra é um vírus bacteriano chamado PBS1 que contém uracila no seu ADN. Em contraste, após a síntese de certas moléculas de ARN, um número significante de uracilas são convertidas a timinas pela adição enzimática do grupo de metila. Isto acontece principalmente em RNAs estruturais e enzimáticos como o ARN mensageiro e o ARN ribossomal.
A dupla hélice é uma espiral dextra. Como as cadeias de ADN giram uma ao redor da outra, elas deixam espaços entre cada cerne de fosfato, revelando os sítios das bases que estão localizadas na parte interna. Há dois destes espaços ao redor da superfície da dupla hélice: um espaço é maior e possui 22 Å de largura e o outro, o espaço é menor com 12 Å de largura. Proteínas como fatores de transcrição podem ligar-se a sequências específicas do ADN de dupla cadeia, normalmente estabelecendo contato com os sítios das bases expostos no espaço maior.
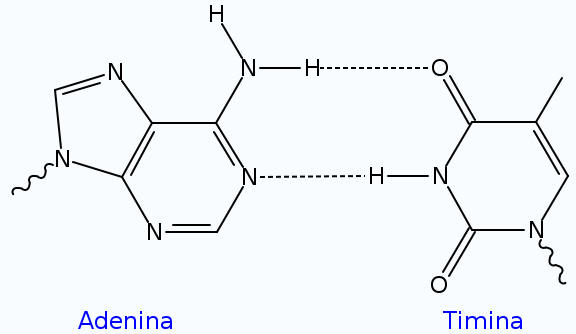
Emparelhamento de bases
Cada tipo de base numa cadeia forma uma ligação com apenas um tipo de base na outra cadeia. Este comportamento é designado de complementariedade de bases. Assim, as purinas formam pontes de hidrogênio com pirimidinas, i.e. A liga-se com T e C com G. Este arranjo de dois nucleotídeos complementares na dupla hélice é chamado par de base. Além das pontes de hidrogênio entre as bases, as duas fitas são mantidas juntas devido a forças geradas por interações hidrofóbicas entre as bases empilhadas, a qual não é influenciada pela sequência do DNA. [12] Como as pontes de hidrogênio não são ligações covalentes, podem ser quebradas e reunidas com relativa facilidade. Desta forma, as duas fitas da dupla hélice de DNA podem ser separadas como um "zíper" (fecho) por força mecânica ou altas temperaturas.[13] Como resultado desta complementariedade, toda a informação contida numa das cadeias de DNA está também contida na outra, o que é fundamental para a replicação do DNA. [5]
Os dois tipos de pares de base formam diferentes números de pontes de hidrogênio: AT forma duas pontes de hidrogênio enquanto que GC formam três pontes de hidrogênio. Desta forma a interação entre GC é mais forte que AT. Como resultado, a percentagem de GC numa dupla fita de DNA determina a força de interação entre as duas cadeias. [14] Uma parte da dupla cadeia de DNA que precisa de ser separada facilmente, tal como a TATAAT Caixa de Pribnow nos promotores bacterianos, tendem a ter as sequências com maior predomínio de AT, para facilitar a abertura da dupla cadeia quando da transcrição. No laboratório, a força desta interação pode ser medida encontrando a temperatura necessária para quebrar as pontes de hidrogênio, a temperatura de desnaturação (também chamado Tm). Quando todos os pares de base numa dupla hélice de ADN quebram as suas ligações, as duas cadeias separam-se e existem em solução como duas moléculas completamente independentes. Estas moléculas de DNA de cadeia simples não têm uma única forma comum, mas algumas conformações são mais estáveis do que outras[15].
Fenda maior e menor
O DNA normalmente encontra-se em forma de uma espiral. Portanto, as fitas de DNA giram uma sobre a outra e acabam por formar fendas entre os cernes de fosfatos, deixando expostas as faces das bases nitrogenadas que não estão unidas por pontes de hidrogênio com a base complementar.
Há dois tipos de fendas na superfície da dupla hélice: uma com 22 Å denominada fenda maior e uma com 12 Å designada de fenda menor. [16] A principal função das “fendas” do DNA é fornecer a informação acerca das bases que se encontram ligadas numa determinada região da dupla cadeia sem a necessidade de a abrir. Como é de esperar, a fenda maior oferece uma maior acessibilidade de ligação com proteínas do que a fenda menor, mas isso não quer dizer que a fenda menor não possa interagir com proteínas. Um exemplo disto é a TBP (TATA-binding protein) uma importante proteína para a transcrição em eucariotas. [17]
Senso e anti-senso
Uma sequência de DNA é chamada de senso se possui a mesma sequência do RNAm. A cadeia oposta (complementar) à cadeia "senso" é denominada sequência anti-senso. Como a RNA polimerase sintetiza um RNA que é complementar à fita molde, então podemos dizer que ela utiliza a cadeia anti-senso como molde para produzir um RNA. As sequências senso e anti-senso podem existir em diferentes partes da mesma cadeia de DNA, que pode ser de um lado ou do outro, dependendo de onde se encontra a sequência codificadora.
Às vezes não é possível dizer qual é a cadeia senso ou anti-senso. Isto acontece devido à existência de genes que se sobrepõem, e neste caso ambas as cadeias dão origem a um RNA. [18] Nas bactérias, a sobreposição pode estar envolvida da regulação da transcrição. [19] Já nos vírus, a sobreposição aumenta a capacidade do armazenamento de informações em pequenos genomas virais. [20]
Supercoiling (super-helicoidização)
O DNA pode ser torcido num processo denominado super-helicoidização. No estado relaxado do DNA, uma fita normalmente dá uma volta completa ao eixo da dupla hélice a cada 10,4 pares de base, mas se o DNA está torcido, as cadeias ficam mais ou menos enroladas. [21] Se o DNA está torcido na direção da hélice, é denominado um supercoiling positivo e as bases estão unidas mais firmemente. Já o supercoiling negativo refere-se a uma torção na direção oposta resultanto num afrouxamento das bases. Na natureza, o DNA apresenta um ligeiro supercoiling negativo que é causado pela ação de uma enzima denominada topoisomerase. [22] Estas enzimas também são necessárias para aliviar o estresse de torção causado no DNA durante os processos de transcrição e replicação. [23]
Estrutura alternativa da dupla hélice
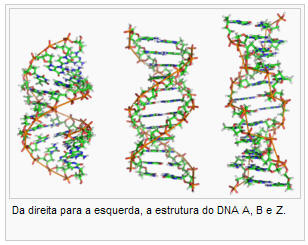
O DNA pode existir em muitas formações diferentes. As formações mais comuns são: DNA-A, DNA-B, DNA-C, DNA-D, [24] DNA-E,[25] DNA-H,[26] DNA-L,[24] DNA-P,[27] e DNA-Z.[28] Porém, só as formações de DNA A, B e Z foram encontradas em sistemas biológicos naturais. A formação que o DNA adopta depende de vários fatores da própria sequência de DNA: a intensidade e direção do supercoiling, modificações químicas das bases e a solução na qual o DNA está presente (ex.: concentração de metais, íons e poliaminas). [29] Das três formações referidas, a forma “B” é a mais comum nas condições encontradas nas células. [30]
A forma “A” corresponde à espiral dextra mais larga, com uma fenda menor larga e superficial e uma fenda maior estreita e profunda. A forma “A” ocorre sob condições não fisiológicas em amostras de DNA desidratadas, enquanto na célula pode ser produzida por pareamento híbrido de DNA e RNA ou pelo complexo enzima-DNA. [31][32] Em segmentos de DNA onde as bases foram quimicamente modificadas por metilação, o DNA pode sofrer uma grande modificação na sua formação e adoptar a forma DNA-Z. Aqui, a cadeia gira sobre o eixo da dupla hélice para a esquerda, o oposto da forma mais comum – DNA-B. [33] Esta estrutura é rara e pode ser reconhecida por proteínas especificas de ligação com o DNA-Z e podem estar envolvidas na regulação da transcrição. [34]
Estruturas em quadruplex
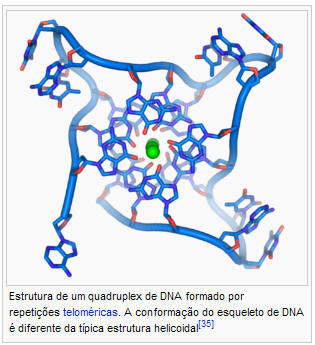
Nas extremidades do cromossomas lineares estão zonas especializadas do DNA chamadas telômeros. A função principal destas regiões é permitir que a célula replique as extremidades do cromossoma usando a enzima telomerase, porque enzimas que permitem replicar DNA normalmente não conseguem copiar as extremidades 3' dos cromossomas.[36] Estas tampas de cromossoma especializadas também ajudam a proteger as extremidades do DNA, e evitam que o sistema de reparo de DNA da célula as trate como danos que precisassem de ser corrigidos.[37] Em células humanas, os telômeros tem normalmente vários milhares de repetições de uma sequência simples (TTAGGG).[38]
Estas sequências ricas em guanina podem estabilizar as extremidades dos cromossomas formando estruturas de unidades de quatro bases empilhadas, ao invés dos pares de base usuais encontrados em outras moléculas de DNA. Aqui, quatro bases de guanina formam uma placa chata e depois estas unidade chatas de quatro bases empilham-se no topo umas das outras, para formarem estruturas G-quadruplex estáveis.[39] Estas estruturas são estabilizadas por pontes de hidrogênio entre as margens das bases e por quelação de um íon metálico no centro de cada unidade de quatro-bases.[40] Outras estruturas podem também ser formadas, com o conjunto central de quatro bases a vir quer de uma cadeia simples enrolada à volta das bases ou de diversas cadeias paralelas, cada uma contribuindo com uma base para a estrutura central.
Além destas estruturas empilhadas, os telómeros também forma grandes estruturas em forma de laço chamados telomere loops ou T-loops. Aqui, a DNA de cadeia simples enrola-se à volta de um círculo grande estabilizado por proteínas que se ligam a telómeros.[41] Mesmo no fim dos T-loops, o DNA de cadeia simples do telómero é segurado sobre uma região de DNA de cadeia dupla pela cadeia do telómero que desestabiliza o DNA de dupla hélica e o emparelhamento de bases de uma das duas cadeias. Esta estrutura de cadeia tripla é chamada de laço de deslocamento ou D-loop.[39]
Modificações químicas
|
citosina |
5-metilcitosina |
timina |
Estrutura da citosina com e sem o grupo 5-metil.
Depois de deaminação, a 5-metilcitosina tem a mesma
estrutura da timina
Modificações de bases
A expressão de genes é influenciado pela maneira que o DNA está arrumado nos cromossomas, numa estrutura chamada cromatina. As modificações de bases podem estar envolvidas na arrumação, com as regiões quem tem expressão génica baixa ou inexistente contendo usualmente níveis elevados de metilação das bases de citosina. Por exemplo, a metilação de citosina produz 5-metilcitosina, que é importante na inativação do cromossoma X.[42] O nível médio de metilação varia entre organismos - o verme Caenorhabditis elegans tem pouca metilação da citosina, enquanto que vertebrados têm níveis mais elevados, com até 1% do seu DNA contendo 5-metilcitosina[43] Apesar da importância da 5-metilcitosina, esta pode desaminar transformando-se em timina. Citosinas metiladas são por isso especialmente susceptíveis de sofrer mutações.[44] Outras modificações de bases incluem metilação de adeninas em bactérias e glicolisação do uracilo para produzir a "base-J" em organismos da classe Kinetoplastea.[45][46]
Danificação do DNA
O DNA pode ser danificado por muitos tipos diferentes de mutagênicos, que mudam a sequência de DNA. Mutagênicos incluem agentes oxidantes, agentes alquilantes e também por radiação eletromagnéticas de grande energia tal como luz ultravioleta e raios-X. O tipo de danificação de DNA produzido depende do tipo de mutagênico. A luz ultravioleta, por exemplo, pode danificar o DNA produzindo dímeros de timina, que são ligações cruzadas entre pirimidinas.[48] Por outro lado, oxidantes como radicais livres ou peróxido de hidrogênio produzem múltiplos tipos de danos, incluindo modificações de bases, em particular guanosina, e quebras das cadeias duplas.[49] Em cada célula humana, cerca de 500 bases sofrer danos por oxidação por dia.[50][51] De entre estas lesões oxidativas, a mais perigos são as quebras da cadeia dupla, pois estas são difíceis de reparar e podem produzir mutações pontuais, inserções e deleções, assim como translocações cromossômicas.[52]
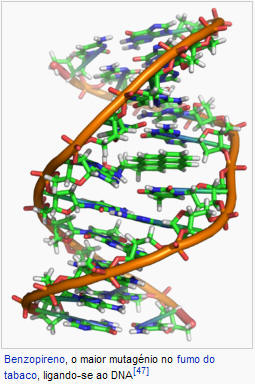
Muitos mutagénios encaixam entre o espaço entre dois pares de base adjacentes, a chamada intercalação. A maioria dos intercaladores são aromáticos e moléculas planas e incluem brometo de etídio, daunomicina, doxorrubicina e talidomida. Para que um intercalador encaixe entre pares de bases, as bases tem de se separar, distorcendo as cadeias de DNA, desenrolando a cadeia dupla. Isto inibe quer a transcrição como a replicação do DNA, causando toxicidade e mutações. Como resultado, os intercaladores de DNA são muitas vezes carcinogênicos. Benzopireno, acridinas, aflatoxina e brometo de etídio são exemplos bem conhecidos.[53][54][55] No entanto, devido à sua capacidade de inibir a transcrição e replicação, estas toxinas também são usadas em quimioterapia para inibir células de cancro com crescimento rápido.[56]
Funções biológicas
O DNA ocorre normalmente como cromossomas lineares em eucariotas, e como cromossomas circulares em procariotas. O conjunto dos cromossomas numa célula perfazem o seu genoma; o genoma humano tem aproximadamente 3 mil milhões de pares de base dispostos em 46 cromossomas[57]. A informação transportada pelo DNA está contida nas sequências de pedaços de DNA chamados genes. A transmissão da informação genética dos genes é conseguida via a complementaridade do emparelhamento das bases. Por exemplo, na transcrição, quando uma célula usa a informação num gene, a sequência de DNA é copiado para uma sequência de RNA complementar através da atração entre o DNA e os nucleotídeos de RNA corretos. Normalmente, esta cópia de RNA é depois usada para fazer uma sequência protéica correspondente no processo de tradução que depende da mesma interação entre nucleotídeos de RNA. Alternativamente, uma célula pode simplesmente copiar a sua informação genética num processo chamado replicação do DNA.
Genes e genomas
O DNA genômico está localizado no núcleo celular dos eucariontes, assim como em pequenas quantidades em mitocôndrias e em cloroplastos. Em procariontes, o DNA está dentro de um corpo de forma irregular no citoplasma chamado nucleóide[58] A informação genética num genoma está nos genes, e o conjunto completo desta informação num organismo é chamado o seu genótipo. Um gene é a unidade básica da hereditariedade e é uma região do DNA que influencia uma característica particular num organismo. Genes contêm uma open reading frame que pode ser transcrita, assim como sequências reguladoras tais como promotores ou enhancers, que controlam a transcrição da open reading frame.
Em muitas espécies, apenas uma pequena fração da sequência total do genoma codifica uma proteína. Por exemplo, apenas 1,5% do genoma humano consiste de exões (que codificam proteínas), com mais de 50% do DNA humano consistindo de sequências repetitivas[59]. As razões para a presença de tanto DNA não-codificante em genomas eucarióticos e as extraordinárias diferenças no tamanho do genoma, ou valor C, entre espécies representam um enigma ainda não decifrado conhecido por "C-value enigma" (paradoxo do valor C)[60]. Contudo, sequências de DNA que não codificam proteínas podem ainda codificar moléculas de RNA não-codificante funcional, que estão envolvidas na regulação da expressão génica[61].

Algumas sequências de DNA não-codificante tem um papel estrutural nos cromossomas. Os telômeros e centrômeros contêm tipicamente poucos genes, mas são importantes para a função e estabilidade dos cromossomas[37][63]. Uma forma abundante de DNA não codificante em humanos são pseudogenes, que são cópias de genes que foram desabilitados por mutação[64]. Estas sequências são usualmente apenas fósseis moleculares, apesar de poderem servir ocasionalmente como material genético em bruto para a criação de novos genes através do processo de duplicação de genes e divergência[65].
Transcrição e tradução
Um gene é uma sequência de DNA que contêm informação genética e pode influenciar o fenótipo de um organismo. Dentro de um gene, a sequência de bases ao longo de uma cadeia de DNA definem uma cadeia de RNA mensageiro, que por sua vez define uma ou mais sequências protéicas. A relação entre a sequência de nucleotídeos de um gene e a sequência de aminoácidos de uma proteína é determinada pelas regras de tradução, conhecidas coletivamente como o código genético. O código genético consiste de 'palavras' de três letras chamadas códons formadas por uma sequência de três nucleotídeos (p.e. ACU, CAG, UUU).
Na transcrição, os códons de um gene são copiados para um RNA mensageiro pela RNA polimerase. Esta cópia de RNA é depois descodificada por uma ribossoma que lê a sequência de RNA emparelhando o RNA mensageiro a RNA de transferência, que carrega aminoácidos. Uma vez que há quatro bases em combinações de 3-letras, há 64 códons possíveis (43 combinações). Estas codificam os vinte aminoácidos, dando à maioria dos aminoácidos mais do que um códon possível. Há também três códons 'stop' ou 'nonsense' significando o fim da região codificante; estes são os códons UAA, UGA e UAG.
Replicação de DNA. A dupla hélice é desdobrada por uma helicase e por uma topoisomerase. Em seguida, uma DNA polimerase produz uma cópia da cadeia líder. Outra DNA polimerase liga-se à cadeia atrasada. Esta enzima produz segmentos descontínuos (chamados fragmentos de Okazaki) antes que a DNA ligase os juntar
Replicação
A divisão celular é essencial para que um organismo cresça, mas quando uma célula se divide tem de replicar o DNA do seu genoma para que as duas células-filha tenham a mesma informação genética que a célula parental. A estrutura em dupla-hélice do DNA fornece um mecanismo simples para a sua replicação. As duas cadeias são separadas e depois sequências de DNA, complementares a cada uma das cadeias são recreadas por uma enzima chamada DNA polimerase. Esta enzima constrói a cadeia complementar encontrando a base correta através de emparelhamento com a base complementar, e ligando-a à cadeia original. Como as polimerases de DNA só conseguem fazer a extensão de uma cadeia de DNA na direção 5' para 3', outros mecanismos são usados para copiar a cadeia antiparalela da dupla hélice.[66] Desta forma, a base presente na cadeia antiga dita que base vai aparecer na nova cadeia e a célula acaba com uma cópia perfeita do seu DNA.
Interações com proteínas
Todas as funções do DNA dependem de interações com proteínas. Estas interações com proteínas podem ser não-específicas, ou a proteína pode ligar-se especificamente a uma única sequência de DNA. Algumas enzimas também se podem ligar ao DNA. Destas, as polimerases que copiam as sequências de DNA na transcrição e replicação são particularmente importantes.
Proteínas que se ligam ao DNA (DNA-binding)
Interação do DNA com histonas (mostrado em branco, em cima). Os aminoácidos básicos destas proteínas (em baixo à esquerda, em azul) liga-se aos grupos fosfato do DNA (em baixo à direita, em vermelho)
Proteínas estruturais que se ligam ao DNA são exemplos bem estudados de interações não-específicas DNA-proteínas. Nos cromossomas, o DNA está ligado a proteínas estruturais formando complexos. Estas proteínas organizam o DNA numa estrutura compacta, a cromatina. Em eucariontes esta estrutura envolve a ligação do DNA a um complexo de pequenas proteínas básicas chamadas histonas, enquanto que em procariontes estão envolvidas vários tipos de proteínas.[67][68] As histonas formam um complexo em forma de disco, o nucleossoma, que contem duas voltas completas de DNA de cadeia dupla enrolado à sua volta. Estas interações não específicas formam-se quando os resíduos básicos das histonas fazem ligações iônicas ao esqueleto açúcar-fosfato acídico do DNA, e por isso são largamente independentes da sequência de bases.[69] Modificações químicas nestes resíduos de aminoácidos inclui metilação, fosforilação e acetilação.[70] Estas mudanças químicas alteram a força da interação entre o DNA e as histonas, tornando o DNA mais ou menos acessível a fatores de transcrição e mudando a taxa de transcrição.[71] Outras proteínas com ligação a DNA não-específicas incluem o grupo de proteínas de alta mobilidade, que se ligam a DNA dobrado ou distorcido.[72] Estas proteínas são importantes pois dobram conjuntos de nucleossomas e organizam-nos em estruturas maiores que perfazem os cromossomas.[73]
Um grupo distinto destas proteínas são as que se ligam especificamente a DNA de cadeia simples. Nos humanos, a proteína de replicação A é o membro desta família melhor compreendido e é usado em processos onde a dupla hélice é separada, incluindo durante a replicação do DNA, recombinação e reparo.[74] Estas proteínas parecem estabilizar DNA de cadeia dupla e protegem-no da formação de hairpin loops ou de ser degradado por nucleases.
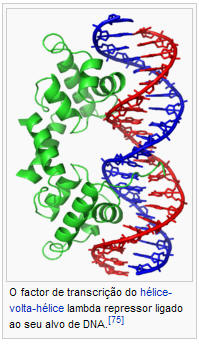
Em contraste, outras proteínas evoluiram de modo a ligar-se a sequências de DNA específicas. Os fatores de transcrição são dos mais intensivamente estudados, que são proteínas que regulam a transcrição. Cada fator de transcrição liga-se a um conjunto particular de sequências de DNA e ativa ou inibe a transcrição de genes que tenham estas sequências perto dos seus promotores. Os fatores de transcrição fazem isto de duas maneiras. Primeiro, podem ligar-se à polimerase do RNA responsável pela transcrição, quer diretamente quer através de proteínas mediadoras; isto posiciona a polimerase no promotor e permite que comece a transcrição.[76] Em alternativa, os fatores de transcrição podem ligar-se a enzimas que modificam as histonas no promotor; isto muda a acessibilidade do molde de DNA à polimerase.[77]
Como estes locais de ligação podem ocorrer pelo genoma inteiro de um organismo, mudanças na atividade de um tipo de fator de transcrição pode afetar milhares de genes.[78] Por consequência, estas proteínas são muitas vezes alvo de processos de transdução de sinal que controlam respostas a mudanças ambientais ou diferenciação e desenvolvimento celular. A especificidade da interação destes factores de transcrição com o DNA provem das proteínas que fazem contactos múltiplos com a beira das bases de DNA, permitindo a "leitura" da sequência de DNA, A maior parte destas interações com bases fazem-se na fenda maior, onde as bases estão mais acessíveis.[79]
Enzimas que modificam o ADN
Nucleases e ligases
As nucleases são enzimas que cortam as cadeias de ADN mediante a catálise da hidrólise das ligações fosfodiéster. As nucleases que hidrolizam nucleotídeos a partir dos extremos das cadeias de ADN denominam-se exonucleases, enquanto que as endonucleases cortam no interior das cadeias. As nucleases que se utilizam com maior frequência em biologia molecular são as enzimas de restrição, endonucleases que cortam o ADN em sequências específicas. Por exemplo, a enzima EcoRV, mostrada à esquerda, reconhece a sequência de 6 bases 5′-GAT|ATC-3′ e faz um corte em ambas as cadeias na linha vertical indicada, gerando duas moléculas de ADN. Outras enzimas de restrição geram, no entanto, extremidades coesivas, já que cortam de forma diferente as duas cadeias de ADN. Na natureza, estas enzimas protegem as bactérias contra as infecções de fagos, ao digerir o ADN do fago quando entra através da parede bacteriana, atuando como um mecanismo de defesa.[81] Em biotecnologia, estas nucleases específicas utilizam-se na clonagem molecular e na técnica de impressão de ADN (DNA fingerprinting, em inglês).
As enzimas denominadas DNA ligases podem reunir pedaços de ADN cortados ou quebrados.[82] As ligases são particularmente importantes na replicação do ADN da cadeia atrasada de ADN, já que unem os fragmentos curtos de ADN generados no garfo de replicação para formar uma copia completa do molde de ADN. Também se utilizam no reparo de ADN e na recombinação genética.[82]
Topoisomerases e helicases
As topoisomerases são enzimas que possuem a atividade nuclease e ligase. Estas proteínas mudam a quantidade de ADN superenrolado. Algumas destas enzimas funcionam cortando a hélice de ADN e permitindo a uma secção que faça rotação, de maneira a reduzir o grau de superenrolamento; uma vez feito isto, a enzima volta a unir os fragmentos de ADN.[22] Outros tipos de enzimas são capazes de cortar uma hélice de ADN e depois passar a segunda cadeia de ADN através desta quebra, antes de reunir as hélices.[83] As topoisomerases são necessárias para muitos processos em que intervém o ADN, como a replicação do ADN e a transcrição.[23]
As helicases são proteínas que pertencem ao grupo dos motores moleculares. Utilizam energia química armazenada nos trifosfatos de nucleósidos, fundamentalmente ATP, para romper pontes de hidrogênio entre bases e separar a dupla hélice de ADN em cadeias simples.[84] Estas enzimas são essenciais para a maioria dos processos em que as enzimas necessitam de aceder às bases do ADN.
Polimerases
As polimerases são enzimas que sintetizam cadeias de nucleotídeos a partir de trifosfatos de nucleósidos. A sequência de seus produtos são cópias de cadeias de polinucleotídeos existentes, que se denominam moldes. Estas enzimas funcionam adicionando nucleotídeos ao grupo hidróxido em 3' do nucleotídeo anterior numa cadeia de ADN. Por consequência, todas as polimerasas funcionam na direção 5′ --> 3′.[85] Nos sítios ativos destas enzimas, o trifosfato de nucleósido que se incorpora emparelha a sua base com a correspondente no molde: isto permite que a polimerase sintetize de forma precisa a cadeia complementar ao molde.
As polimerases classificam-se de acordo com o tipo de molde que utilizam:
|
Na replicação do ADN, uma ADN polimerase dependente de ADN realiza uma cópia de ADN a partir de uma sequência de ADN. A precisão é vital neste processo, por isso muitas destas polimerases possuem uma atividade de verificação de leitura (proofreading). Mediante esta atividade, a polimerase reconhece erros ocasionais na reação de síntese, devido à falta de emparelhamento entre o nucleotídeo errôneo e o molde, o que gera um desacoplamento (mismatch). Se se detecta um desacoplamento, ativa-se uma atividade exonuclease na direção 3′ --> 5′ e a base incorreta é eliminada.[86] Na maioria dos organismos, as ADN polimerases funcionam num grande complexo denominado replissoma, que contém múltiplas unidades acessórias, como helicases.[87] |
|
As ADN polimerases dependentes de ARN são uma classe especializada de polimerases que copiam a sequência de uma cadeia de ARN em ADN. Incluem a transcriptase reversa, que é uma enzima viral implicada na infecção de células por retrovírus, e a telomerase, que é necessária para a replicação dos telómeros.[88][36] A telomerase é uma polimerase inusual, porque contém o seu próprio molde de ARN como parte da sua estrutura.[37] |
|
A transcrição é levada a cabo por uma ARN polimerase dependente de ADN que copia a sequência de uma das cadeias de ADN em ARN. Para começar a transcrever um gene, a ARN polimerase une-se a uma sequência do ADN denominada promotor, e separa as cadeias de ADN. Então copia a sequência do gene num transcrito de ARN mensageiro até que alcança uma região do ADN denominada terminador, onde se detém e se separa do ADN. Como ocorre com as ADN polimerases dependentes de ADN em humanos, a ARN polimerase II (a enzima que transcreve a maioria dos genes do genoma humano) funciona como um grande complexo multiproteico que contém múltiplas subunidades reguladoras e acessórias.[89] |
Recombinação genética
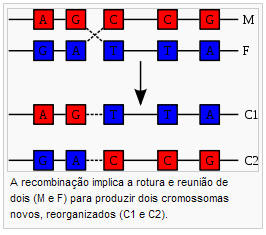
Uma hélice de ADN normalmente não interage com outros segmentos de ADN, e nas células humanas os diferentes cromossomas ocupam áreas separadas no núcleo celular denominadas “territórios cromossômicos”.[91] A separação física dos diferentes cromossomas é importante para que o ADN mantenha a sua capacidade de funcionar como um armazém estável de informação. Um dos poucos momentos em que os cromossomas interagem é durante o sobrecruzamento cromossômico (chromosomal crossover, em inglês), durante o qual se recombinam. O sobrecruzamento cromossômico ocorre quando duas hélices de ADN se rompem, sofrem intercâmbio e se unem novamente.
A recombinação permite aos cromossomas trocar informação genética e produzir novas combinações de genes, o que aumenta a eficiência da seleção natural e pode ser importante na evolução rápida de novas proteínas.[92] Durante a profase I da meiose, uma vez que os cromossomas homólogos estão perfeitamente emparelhados formando estruturas que se denominam bivalentes, produz-se o fenômeno de sobrecruzamento ou entrecruzamento (crossing-over), no qual os cromatídeos homólogas não irmãos (procedentes do pai e da mãe) trocam material genético. A recombinação genética resultante faz aumentar em grande medida a variação genética entre a descendência de progenitores que se reproduzem por via sexual. A recombinação genética também pode estar implicada na reparação do ADN, em particular na resposta celular às roturas da dupla cadeia (double-strand breaks).[93]
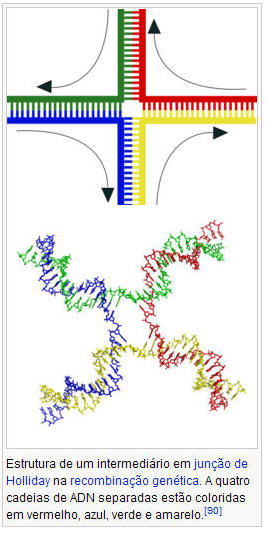
A forma mais frequente de sobrecruzamento cromossômico é a recombinação homóloga, na qual os dois cromossomas implicados compartilham sequências muito similares. A recombinação não-homóloga pode ser danosa para as células, já que pode produzir translocações cromossômicas e anormalidades genéticas. A reação de recombinação é catalisada por enzimas conhecidas como recombinases, tais como a RAD51.[94] O primeiro passo no processo de recombinação é uma rotura da dupla cadeia, causada por uma endonuclease ou por dano no ADN.[95] Posteriormente, uma série de passos catalisados em parte pela recombinase conduz à união das duas hélices formando pelo menos uma junção de Holliday, na qual um segmento de uma cadeia simples é anelada com a cadeia complementar na outra hélice. A junção de Holliday é uma estrutura de união tetraédrica que pode mover-se ao longo do par de cromossomas, intercambiando uma cadeia por outra. A reação de recombinação detém-se pelo corte da união e a reunião dos segmentos de ADN libertados.[96]
Evolução do metabolismo de ADN
O ADN contém a informação genética que permite à maioria dos organismos vivos funcionar, crescer e reproduzirem-se. No entanto, não é claro durante quanto tempo exerceu esta função nos ~3000 milhões de anos desde a história da vida, já que se propôs que as formas de vida mais precoces poderiam ter utilizado ARN como material genético.[85][97] O ARN poderia ter funcionado como parte central de um metabolismo primogênito, já que pode transmitir informação genética e simultaneamente atuar como catalisador, formando parte das ribozimas.[98] Este antigo mundo de ARN onde os ácidos nucléicos funcionariam como catalisadores e como armazéns de informação genética poderia ter influenciado na evolução do código genético atual, baseado em quatro nucleotídeos. Isto se deveria a que o número de bases únicas num organismo é um compromisso entre um número pequeno de bases (o que aumentaria a precisão da replicação) e um número grande de bases (que por sua vez aumentaria a eficiência catalítica das ribozimas).[99]
Infelizmente, não dispomos de evidência direta dos sistemas genéticos ancestrais, porque a recuperação do ADN a partir da maior parte dos fósseis é impossível. Isto se deve a que o ADN é capaz de sobreviver no meio ambiente durante menos de um milhão de anos, e logo começa a degradar-se lentamente em fragmentos de menor tamanho em solução.[100] Algumas investigações pretendem a obtenção de ADN mais antigo, por exemplo o isolamento de uma bactéria viável a partir de um cristal salino de 250 milhões de anos de antiguidade,[101] mas estes dados são controversos.[102][103]
No entanto, podem utilizar-se ferramentas de evolução molecular para inferir os genomas de organismos ancestrais a partir de organismos contemporâneos.[104][105] Em muitos casos, estas inferências são suficientemente fiáveis, de maneira que uma biomolécula codificada num genoma ancestral pode ser ressuscitada no laboratório para ser estudada hoje.[106][107] Uma vez que a biomolécula ancestral foi ressuscitada, as suas propriedades podem oferecer inferências sobre ambientes e estilos de vida primigénios. Este processo relaciona-se com o campo emergente da paleogenética experimental.[108] Apesar de tudo, o processo de trabalho até atrás desde o presente tem limitações inerentes, razão pela qual outros investigadores tratam de elucidar o mecanismo evolutivo trabalhando desde a origem da Terra até adiante no tempo. Dada suficiente informação sobre a química no cosmos, como as substâncias cósmicas poderiam haver-se depositado na Terra, e as transformações que poderiam ter tido lugar na superfície terrestre primigénia, talvez poderíamos ser capazes de aprender sobre as origens para desenvolver modelos de evolução da informação genética até diante no tempo.
Aplicações
Engenharia genética
A investigação sobre o ADN tem um impacto significativo, especialmente no âmbito da medicina, mas também na agricultura e criação de gado (onde os objetivos são os mesmos que com as técnicas tradicionais que o homem utiliza desde há milênios - a domesticação, a seleção e os cruzamentos dirigidos - para obter raças de animais e plantas mais produtivos). A moderna biologia e bioquímica fazem uso intensivo da tecnologia do ADN recombinante, introduzindo genes de interesse em organismos, com o objetivo de expressar uma proteína recombinante concreta, que pode ser:
|
isolada para seu uso posterior: por exemplo, podem-se transformar microorganismos para os converter em autênticas fábricas que produzem grandes quantidades de substâncias úteis, como a insulina, que posteriormente se isolam e se utilizam em terapias.[109][110][111] |
|
necessária para substituir a expressão de um gene endógeno danificado que dê lugar a uma patologia, o que permitiria o restabelecimento da atividade da proteína perdida e eventualmente a recuperação do estado fisiológico normal, não patológico. Este é o objetivo da terapia genética, um dos campos em que se está a trabalhar ativamente em medicina, analisando vantagens e inconvenientes de diferentes sistemas de administração do gene (virais e não virais) e os mecanismos de seleção do ponto de integração dos elementos genéticos no genoma alvo.[112] Neste caso, antes de apresentar-se a possibilidade de realizar uma terapia gênica numa determinada patologia, é fundamental compreender o impacto do gene de interesse no desenvolvimento de dita patologia, para o qual é necessário o desenvolvimento de um modelo animal, eliminando ou modificando dito gene num animal de laboratório, mediante a técnica knockout.[113] Só no caso de os resultados no modelo animal sejam satisfatórios se procederia a analisar a possibilidade de restabelecer o gene danificado mediante terapia gênica. |
|
utilizada para enriquecer um alimento: por exemplo, a composição do leite (que é uma importante fonte de proteínas para o consumo humano e animal) pode modificar-se mediante transgenese, adicionando genes exógenos e inativando genes endógenos para melhorar o seu valor nutricional, reduzir infecções nas glândulas mamárias, proporcionar aos consumidores proteínas antipatogênicas e preparar proteínas recombinantes para o uso farmacêutico.[114][115] |
|
útil para melhorar a resistência do organismo transformado: por exemplo, em plantas podem-se introduzir genes que conferem resistência a agentes patogênicos (vírus, insetos, fungos), assim como a agentes estressantes abióticos (salinidade, seca, metais pesados).[116][117][118] |
Medicina forense
Os médicos forenses podem utilizar o ADN presente no sangue, no sêmen, na pele, na saliva ou em pelos, existentes na cena de um crime, para identificar o responsável. Esta técnica denomina-se impressão genética, ou também perfil de ADN. Ao realizar a impressão genética, compara-se o comprimento de secções altamente variáveis do ADN repetitivo, como os micros satélites, entre pessoas diferentes. Este método é frequentemente muito fiável para identificar um criminoso.[119] No entanto, a identificação pode complicar-se se a cena do crime estiver contaminada com ADN de pessoas diferentes.[120] A técnica da impressão genética foi desenvolvida em 1984 pelo geneticista britânico Sir Alec Jeffreys,[121] e utilizada pela primeira vez em medicina forense para condenar Colin Pitchfork por causa dos assassinatos de Narborough (Reino Unido) em 1983 e 1986.[122] Pode-se requerer às pessoas acusadas de certos tipos de crimes que proporcionem una amostra de ADN para ser introduzida numa base de dados. Isto tem facilitado o trabalho dos investigadores na resolução de casos antigos, onde só se obteve uma amostra de ADN da cena do crime, em alguns casos permitindo exonerar um convicto. A impressão genética também pode ser utilizado para identificar vítimas de acidentes em massa,[123] ou para realizar provas de consanguinidade.[124]
Bioinformática
A bioinformática implica a manipulação, busca e extração de informação dos dados da sequência do ADN. O desenvolvimento das técnicas para armazenar e procurar sequências de ADN gerou avanços no desenvolvimento de software para computadores, para muitas aplicações, especialmente algoritmos de busca de frases, aprendizagem automática e teorias de bases de dados.[125] A busca de frases ou algoritmos de coincidências, que procuram a ocorrência de uma sequência de letras dentro de uma sequência de letras maior, desenvolveu-se para buscar sequências específicas de nucleotídeos.[126] Em outras aplicações como editores de textos, inclusive algoritmos simples podem funcionar, mas as sequências de ADN podem gerar que estes algoritmos apresentem um comportamento de quase o pior caso, devido ao baixo número de caracteres. O problema relacionado do alinhamento de sequências persegue identificar sequências homólogas e localizar mutações específicas que as diferenciam. Estas técnicas, fundamentalmente o alinhamento múltiplo de sequências, utilizam-se ao estudar as relações filogenéticas e a função das proteínas.[127] As coleções de dados que representam sequências do ADN do tamanho de um genoma, tais como as produzidas pelo Projecto Genoma Humano, são difíceis de usar sem anotações, que marcam a localização dos genes e dos elementos reguladores em cada cromossoma. As regiões de ADN que têm padrões associados com genes que codificam proteínas ou genes codificantes de ARN, podem identificar-se por algoritmos de localização de genes, o que permite aos investigadores predizer a presença de produtos génicos específicos num organismo mesmo antes que se tenha isolado experimentalmente.[128]
Nanotecnologia de ADN
A nanotecnologia de ADN utiliza as propriedades únicas de reconhecimento molecular de ADN e outros ácidos nucléicos para criar complexos ramificados auto-ensamblados com propriedades úteis. Neste caso, o ADN utiliza-se como um material estrutural, mais que como um portador de informação biológica. [129] Isto conduziu à criação de lâminas periódicas de duas dimensões (ambas baseadas em azulejos, assim como usando o método de "ADN origami"), para além de estruturas em três dimensiones com forma de poliedros.
História e antropologia
O ADN armazena mutações com o tempo, que se herdam, e portanto contém informação histórica, de maneira que comparando sequências de ADN, os geneticistas podem inferir a história evolutiva dos organismos, a sua filogenia.[130] O campo da filogenia é uma ferramenta potente na biologia evolutiva. Se se compararem as sequências de ADN dentro de uma espécie, os geneticistas de populações podem conhecer a história de populações particulares. Isto pode-se utilizar numa ampla variedade de estudos, desde ecologia até antropologia; por exemplo, evidência baseada na análise de ADN está a ser utilizada para identificar as Dez Tribos Perdidas de Israel.[131][132] Por outro lado, o ADN també se utiliza para estudar relaciones familiares recentes.
Vida
Cada ser vivo que habita a Terra possui uma codificação diferente de instruções escritas na mesma linguagem no seu ADN. Estas diferenças geram as diferenças orgânicas entre os organismos vivos.
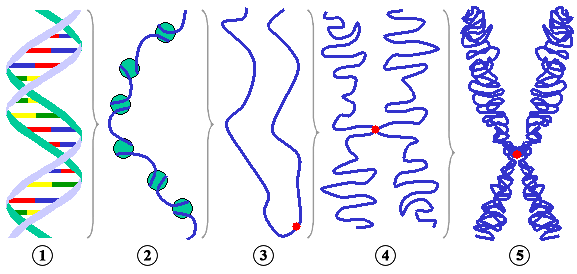
Diferentes níveis de condensação do ADN. (1) Cadeia simples de ADN . (2) Filamento de cromatina (ADN com histonas). (3) Cromatina condensada em intérfase com centrómeros. (4) Cromatina condensada em prófase. (Existem agora duas cópias da molécula de ADN) (5) Cromossoma em metáfase
A dupla cadeia polinucleotídica constitui a molécula de ADN, cuja seqüência de nucleotídeos codifica as instruções hereditárias, organizadas em genes, que codificam as inúmeras proteínas existentes nas mais variadas células. As moléculas de ADN contêm portanto a informação genética necessária para a codificação das características de um indivíduo, como a cor do cabelo em humanos, o formato da folha em Angiospermas e a sua morfologia.
O ADN de todas as células do corpo humano seria equivalente, se fosse visível a olho nu, em comprimento, a oito mil vezes a distância da Terra à Lua.
Criação das moléculas
Erupções vulcânicas seriam comuns na terra primitiva
Presume-se que a Terra, ao se formar de poeira e gases interestelares há aproximadamente 4,6 bilhões de anos, já continha os elementos que posteriormente seriam a base da vida.
Através dos registros fósseis estudados, alguns cientistas afirmam que a vida se desenvolveu em torno de 4 bilhões de anos atrás nos oceanos primitivos do planeta. Segundo alguns, a complexidade das primeiras formas vivas era muito menor que a de qualquer organismo unicelular, que pode ser considerado um ser vivo altamente sofisticado em relação àquelas.
Presume-se que em reações das mais diversas, influenciadas pela luz ultravioleta do Sol, relâmpagos, etc, iniciaram as composições de moléculas bastante simples. Estas eram ricas em hidrogênio procedente da atmosfera primitiva.
Ao avançar do tempo, iniciou-se um processo que levou aqueles fragmentos primitivos a se combinarem e recombinarem, o que gerou moléculas cada vez mais complexas.
Os oceanos da Terra assemelhavam-se a um caldo orgânico porém, ainda não eram vivos. À medida em que a complexidade das moléculas aumentava, começaram a surgir algumas que iniciaram um processo grosseiro de copiarem a si mesmas.
Estas eram provavelmente as primeiras ancestrais do ácido desoxirribonucléico, ou ADN, molécula principal da vida na Terra.
Evolução
Alguns cientistas afirmam que o planeta Terra era um Jardim do Éden molecular, há cerca de quatro mil milhões de anos. As moléculas reproduziam-se de forma ineficiente, produzindo-se cópias grosseiras. Neste tipo de cenário ocorreram as primeiras replicações, mutações, e extinções de forma seletiva e aleatória. As variedades menos eficientes foram sendo eliminadas, enquanto aquelas que conseguiam sobreviver tornaram-se cada vez mais eficientes a cada geração - evolução.
Avançando-se no tempo, as moléculas orgânicas foram adquirindo mais e mais funções especializadas, foram-se juntando aos poucos e de forma casual. A princípio, pode-se dizer que estas coletividades moleculares formaram algo parecido com o primeiro ser vivo composto a partir de algum momento por um ADN funcional.
Bibliografia
|
Introdução à genética, Riffiths, Wessler, Lewontin, Gesbart, suzuki, Miller, 8º Edição, Guanabara Koogan, 2006. | |
|
Biologia; José Mariano Amabis, Gilberto Rodriges Martho; Moderna; 2004 |
-
Dahm R (January 2008). "Discovering DNA: Friedrich Miescher and the early years of nucleic acid research". Hum. Genet. 122 (6): 565–81. doi:10.1007/s00439-007-0433-0. PMID 17901982.
-
# Morange, Michel. A History of Molecular Biology. Cambridge, MA: Harvard University Press. 1998.
-
Lehrer, Steven. Explorers of the Body. 2nd edition 2006 pp 47-52
-
Avery, O T; Macleod C M, McCarty M (October 2000). "Studies on the chemical nature of the substance inducing transformation of pneumococcal types: Induction of transformation by a desoxyribonucleic acid fraction isolated from Pneumococcus type III. Oswald Theodore Avery (1877-1955)". Clin. Orthop. Relat. Res. 379 (379 Suppl): S3–8. doi:10.1097/00003086-200010001-00002. PMID 11039746.
-
5,0 5,1 Alberts, Bruce, Alexander Johnson, Julian Lewis, Kazuo, Martin Raff, Keith Roberts, and Peter Walters (2002). Molecular Biology of the Cell; Fourth Edition. New York and London: Garland Science.
-
Butler, John M. (2001) Forensic DNA Typing "Elsevier". pp. 14–15. ISBN 978-0-12-147951-0.
-
Mandelkern M, Elias J, Eden D, Crothers D (1981). "The dimensions of DNA in solution". J Mol Biol 152 (1): 153–61. PMID 7338906.
-
Gregory S, et al. (2006). "The DNA sequence and biological annotation of human chromosome 1". Nature 441 (7091): 315–21. PMID 16710414.
-
Watson J, Crick F (1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid". Nature 171 (4356): 737–8. PMID 13054692.
-
Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6
-
Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents IUPAC-IUB Commission on Biochemical Nomenclature (CBN) Accessed 03 Jan 2006
-
Ponnuswamy P, Gromiha M (1994). "On the conformational stability of oligonucleotide duplexes and tRNA molecules". J Theor Biol 169 (4): 419–32. PMID 7526075.
-
Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H (2000). "Mechanical stability of single DNA molecules". Biophys J 78 (4): 1997–2007. PMID 10733978.
-
Chalikian T, Völker J, Plum G, Breslauer K (1999). "A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques". Proc Natl Acad Sci U S A 96 (14): 7853–8. PMID 10393911.
-
Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J (2004). "Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern". Biochemistry 43 (51): 15996–6010. PMID 15609994.
-
Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R (1980). "Crystal structure analysis of a complete turn of B-DNA". Nature 287 (5784): 755–8. PMID 7432492.
-
Pabo C, Sauer R (1984). "Protein-DNA recognition". Annu Rev Biochem 53: 293–321. PMID 6236744.
-
Makalowska I, Lin C, Makalowski W (2005). "Overlapping genes in vertebrate genomes". Comput Biol Chem 29 (1): 1–12. PMID 15680581.
-
Johnson Z, Chisholm S (2004). "Properties of overlapping genes are conserved across microbial genomes". Genome Res 14 (11): 2268–72. PMID 15520290.
-
Lamb R, Horvath C (1991). "Diversity of coding strategies in influenza viruses". Trends Genet 7 (8): 261–6. PMID 1771674.
-
Benham C, Mielke S (2005). "DNA mechanics". Annu Rev Biomed Eng 7: 21–53. PMID 16004565.
-
22,0 22,1 Champoux J (2001). "DNA topoisomerases: structure, function, and mechanism". Annu Rev Biochem 70: 369–413. PMID 11395412.
-
23,0 23,1 Wang J (2002). "Cellular roles of DNA topoisomerases: a molecular perspective". Nat Rev Mol Cell Biol 3 (6): 430–40. PMID 12042765.
-
24,0 24,1 Hayashi G, Hagihara M, Nakatani K (2005). "Application of L-DNA as a molecular tag". Nucleic Acids Symp Ser (Oxf) 49: 261–262. PMID 17150733.
-
Vargason JM, Eichman BF, Ho PS (2000). "The extended and eccentric E-DNA structure induced by cytosine methylation or bromination". Nature Structural Biology 7: 758–761. PMID 10966645.
-
Wang G, Vasquez KM (2006). "Non-B DNA structure-induced genetic instability". Mutat Res 598 (1–2): 103–119. PMID 16516932.
-
Allemand, et al (1998). "Stretched and overwound DNA forms a Pauling-like structure with exposed bases". PNAS 24: 14152-14157. PMID 9826669.
-
Palecek E (1991). "Local supercoil-stabilized DNA structures". Critical Reviews in Biochemistry and Molecular Biology 26 (2): 151–226. PMID 1914495.
-
Basu H, Feuerstein B, Zarling D, Shafer R, Marton L (1988). "Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies". J Biomol Struct Dyn 6 (2): 299–309. PMID 2482766.
-
Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (1980). "Polymorphism of DNA double helices". J. Mol. Biol. 143 (1): 49–72. PMID 7441761.
-
Wahl M, Sundaralingam M (1997). "Crystal structures of A-DNA duplexes". Biopolymers 44 (1): 45–63. PMID 9097733.
-
Lu XJ, Shakked Z, Olson WK (2000). "A-form conformational motifs in ligand-bound DNA structures". J. Mol. Biol. 300 (4): 819-40. PMID 10891271.
-
Rothenburg S, Koch-Nolte F, Haag F. "DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles". Immunol Rev 184: 286–98. PMID 12086319.
-
Oh D, Kim Y, Rich A (2002). "Z-DNA-binding proteins can act as potent effectors of gene expression in vivo". Proc. Natl. Acad. Sci. U.S.A. 99 (26): 16666-71. PMID 12486233.
-
Criado a partir de NDB UD0017
-
36,0 36,1 Greider C, Blackburn E (1985). "Identification of a specific telomere terminal transferase activity in Tetrahymena extracts". Cell 43 (2 Pt 1): 405–13. PMID 3907856.
-
37,0 37,1 37,2 Nugent C, Lundblad V (1998). "The telomerase reverse transcriptase: components and regulation". Genes Dev 12 (8): 1073–85. PMID 9553037.
-
Wright W, Tesmer V, Huffman K, Levene S, Shay J (1997). "Normal human chromosomes have long G-rich telomeric overhangs at one end". Genes Dev 11 (21): 2801–9. PMID 9353250.
-
39,0 39,1 Burge S, Parkinson G, Hazel P, Todd A, Neidle S (2006). "Quadruplex DNA: sequence, topology and structure". Nucleic Acids Res 34 (19): 5402–15. DOI:10.1093/nar/gkl655. PMID 17012276.
-
Parkinson G, Lee M, Neidle S (2002). "Crystal structure of parallel quadruplexes from human telomeric DNA". Nature 417 (6891): 876–80. DOI:10.1038/nature755. PMID 12050675.
-
Griffith J, Comeau L, Rosenfield S, Stansel R, Bianchi A, Moss H, de Lange T (1999). "Mammalian telomeres end in a large duplex loop". Cell 97 (4): 503–14. PMID 10338214.
-
Klose R, Bird A (2006). "Genomic DNA methylation: the mark and its mediators". Trends Biochem Sci 31 (2): 89–97. DOI:10.1016/j.tibs.2005.12.008. PMID 16403636.
-
Bird A (2002). "DNA methylation patterns and epigenetic memory". Genes Dev 16 (1): 6–21. DOI:10.1101/gad.947102. PMID 11782440.
-
Walsh C, Xu G (2006). "Cytosine methylation and DNA repair". Curr Top Microbiol Immunol 301: 283–315. DOI:10.1007/3-540-31390-7_11. PMID 16570853.
-
Ratel D, Ravanat J, Berger F, Wion D (2006). "N6-methyladenine: the other methylated base of DNA". Bioessays 28 (3): 309–15. DOI:10.1002/bies.20342. PMID 16479578.
-
Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P (1993). "beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei". Cell 75 (6): 1129–36. DOI:10.1016/0092-8674(93)90322-H. PMID 8261512.
-
Criado a partir de PDB 1JDG
-
Douki T, Reynaud-Angelin A, Cadet J, Sage E (2003). "Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation". Biochemistry 42 (30): 9221–6. DOI:10.1021/bi034593c. PMID 12885257.
-
Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S (1999). "Hydroxyl radicals and DNA base damage". Mutat Res 424 (1–2): 9–21. PMID 10064846.
-
Shigenaga M, Gimeno C, Ames B (1989). "Urinary 8-hydroxy-2′-deoxyguanosine as a biological marker of in vivo oxidative DNA damage". Proc Natl Acad Sci U S A 86 (24): 9697–701. DOI:10.1073/pnas.86.24.9697. PMID 2602371.
-
Cathcart R, Schwiers E, Saul R, Ames B (1984). "Thymine glycol and thymidine glycol in human and rat urine: a possible assay for oxidative DNA damage". Proc Natl Acad Sci U S A 81 (18): 5633–7. DOI:10.1073/pnas.81.18.5633. PMID 6592579.
-
Valerie K, Povirk L (2003). "Regulation and mechanisms of mammalian double-strand break repair". Oncogene 22 (37): 5792–812. DOI:10.1038/sj.onc.1206679. PMID 12947387.
-
Ferguson L, Denny W (1991). "The genetic toxicology of acridines". Mutat Res 258 (2): 123–60. PMID 1881402.
-
Jeffrey A (1985). "DNA modification by chemical carcinogens". Pharmacol Ther 28 (2): 237–72. DOI:10.1016/0163-7258(85)90013-0. PMID 3936066.
-
Stephens T, Bunde C, Fillmore B (2000). "Mechanism of action in thalidomide teratogenesis". Biochem Pharmacol 59 (12): 1489–99. DOI:10.1016/S0006-2952(99)00388-3. PMID 10799645.
-
Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A (2001). "Intercalators as anticancer drugs". Curr Pharm Des 7 (17): 1745–80. DOI:10.2174/1381612013397113. PMID 11562309.
-
Venter J, et al. (2001). "The sequence of the human genome". Science 291 (5507): 1304–51. DOI:10.1126/science.1058040. PMID 11181995.
-
Thanbichler M, Wang S, Shapiro L (2005). "The bacterial nucleoid: a highly organized and dynamic structure". J Cell Biochem 96. DOI:10.1002/jcb.20519. PMID 15988757.
-
Wolfsberg T, McEntyre J, Schuler G (2001). "Guide to the draft human genome". Nature 409 (6822): 824–6. DOI:10.1038/35057000. PMID 11236998.
-
Gregory T (2005). "The C-value enigma in plants and animals: a review of parallels and an appeal for partnership". Ann Bot (Lond) 95 (1): 133–46. DOI:10.1093/aob/mci009. PMID 15596463.
-
The ENCODE Project Consortium (2007). "Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project". Nature 447 (7146): 799–816. DOI:10.1038/nature05874.
-
Criada a partir de PDB 1MSW
-
Pidoux A, Allshire R (2005). "The role of heterochromatin in centromere function". Philos Trans R Soc Lond B Biol Sci 360 (1455): 569–79. DOI:10.1098/rstb.2004.1611. PMID 15905142.
-
Harrison P, Hegyi H, Balasubramanian S, Luscombe N, Bertone P, Echols N, Johnson T, Gerstein M (2002). "Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22". Genome Res 12 (2): 272–80. DOI:10.1101/gr.207102. PMID 11827946.
-
Harrison P, Gerstein M (2002). "Studying genomes through the aeons: protein families, pseudogenes and proteome evolution". J Mol Biol 318 (5): 1155–74. DOI:10.1016/S0022-2836(02)00109-2. PMID 12083509.
-
Albà M (2001). "Replicative DNA polymerases". Genome Biol 2 (1): REVIEWS3002. DOI:10.1186/gb-2001-2-1-reviews3002. PMID 11178285.
-
Sandman K, Pereira S, Reeve J (1998). "Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome". Cell Mol Life Sci 54 (12): 1350–64. DOI:10.1007/s000180050259. PMID 9893710.
-
Dame RT (2005). "The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin". Mol. Microbiol. 56 (4): 858–70. DOI:10.1111/j.1365-2958.2005.04598.x. PMID 15853876.
-
Luger K, Mäder A, Richmond R, Sargent D, Richmond T (1997). "Crystal structure of the nucleosome core particle at 2.8 A resolution". Nature 389 (6648): 251–60. DOI:10.1038/38444. PMID 9305837.
-
Jenuwein T, Allis C (2001). "Translating the histone code". Science 293 (5532): 1074–80. DOI:10.1126/science.1063127. PMID 11498575.
-
Ito T. "Nucleosome assembly and remodelling". Curr Top Microbiol Immunol 274: 1–22. PMID 12596902.
-
Thomas J (2001). "HMG1 and 2: architectural DNA-binding proteins". Biochem Soc Trans 29 (Pt 4): 395–401. DOI:10.1042/BST0290395. PMID 11497996.
-
Grosschedl R, Giese K, Pagel J (1994). "HMG domain proteins: architectural elements in the assembly of nucleoprotein structures". Trends Genet 10 (3): 94–100. DOI:10.1016/0168-9525(94)90232-1. PMID 8178371.
-
Iftode C, Daniely Y, Borowiec J (1999). "Replication protein A (RPA): the eukaryotic SSB". Crit Rev Biochem Mol Biol 34 (3): 141–80. DOI:10.1080/10409239991209255. PMID 10473346.
-
Criado a partir de PDB 1LMB
-
Myers L, Kornberg R (2000). "Mediator of transcriptional regulation". Annu Rev Biochem 69: 729–49. DOI:10.1146/annurev.biochem.69.1.729. PMID 10966474.
-
Spiegelman B, Heinrich R (2004). "Biological control through regulated transcriptional coactivators". Cell 119 (2): 157–67. DOI:10.1016/j.cell.2004.09.037. PMID 15479634.
-
Li Z, Van Calcar S, Qu C, Cavenee W, Zhang M, Ren B (2003). "A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells". Proc Natl Acad Sci USA 100 (14): 8164–9. DOI:10.1073/pnas.1332764100. PMID 12808131.
-
Pabo C, Sauer R (1984). "Protein-DNA recognition". Annu Rev Biochem 53: 293–321. DOI:10.1146/annurev.bi.53.070184.001453. PMID 6236744.
-
Criado a partir de PDB 1RVA
-
Bickle T, Krüger D (1993). "Biology of DNA restriction". Microbiol Rev 57 (2): 434–50. PMID 8336674.
-
82,0 82,1 Doherty A, Suh S (2000). "Structural and mechanistic conservation in DNA ligases.". Nucleic Acids Res 28 (21): 4051–8. DOI:10.1093/nar/28.21.4051. PMID 11058099.
-
Schoeffler A, Berger J (2005). "Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism". Biochem Soc Trans 33 (Pt 6): 1465–70. DOI:10.1042/BST20051465. PMID 16246147.
-
Tuteja N, Tuteja R (2004). "Unraveling DNA helicases. Motif, structure, mechanism and function". Eur J Biochem 271 (10): 1849–63. DOI:10.1111/j.1432-1033.2004.04094.x. PMID 15128295.
-
85,0 85,1 Joyce C, Steitz T (1995). "Polymerase structures and function: variations on a theme?". J Bacteriol 177 (22): 6321–9. PMID 7592405.
-
Hubscher U, Maga G, Spadari S (2002). "Eukaryotic DNA polymerases". Annu Rev Biochem 71: 133–63. DOI:10.1146/annurev.biochem.71.090501.150041. PMID 12045093.
-
Johnson A, O'Donnell M (2005). "Cellular DNA replicases: components and dynamics at the replication fork". Annu Rev Biochem 74: 283–315. DOI:10.1146/annurev.biochem.73.011303.073859. PMID 15952889.
-
Tarrago-Litvak L, Andréola M, Nevinsky G, Sarih-Cottin L, Litvak S (1994). "The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention". FASEB J 8 (8): 497–503. PMID 7514143.
-
Martinez E (2002). "Multi-protein complexes in eukaryotic gene transcription". Plant Mol Biol 50 (6): 925–47. DOI:10.1023/A:1021258713850. PMID 12516863.
-
Criado a partir de PDB 1M6G
-
Cremer T, Cremer C (2001). "Chromosome territories, nuclear architecture and gene regulation in mammalian cells". Nat Rev Genet 2 (4): 292–301. DOI:10.1038/35066075. PMID 11283701.
-
Pál C, Papp B, Lercher M (2006). "An integrated view of protein evolution". Nat Rev Genet 7 (5): 337–48. DOI:10.1038/nrg1838. PMID 16619049.
-
O'Driscoll M, Jeggo P (2006). "The role of double-strand break repair - insights from human genetics". Nat Rev Genet 7 (1): 45–54. DOI:10.1038/nrg1746. PMID 16369571.
-
Vispé S, Defais M (1997). "Mammalian Rad51 protein: a RecA homologue with pleiotropic functions". Biochimie 79 (9-10): 587–92. DOI:10.1016/S0300-9084(97)82007-X. PMID 9466696.
-
{cite journal |author=Neale MJ, Keeney S |title=Clarifying the mechanics of DNA strand exchange in meiotic recombination |journal=Nature |volume=442 |issue=7099 |pages=153–8 |year=2006 |pmid=16838012 | doi = 10.1038/nature04885}}
-
Dickman M, Ingleston S, Sedelnikova S, Rafferty J, Lloyd R, Grasby J, Hornby D (2002). "The RuvABC resolvasome". Eur J Biochem 269 (22): 5492–501. DOI:10.1046/j.1432-1033.2002.03250.x. PMID 12423347.
-
Orgel L. "Prebiotic chemistry and the origin of the RNA world". Crit Rev Biochem Mol Biol 39 (2): 99–123. DOI:10.1080/10409230490460765. PMID 15217990.
-
Davenport R (2001). "Ribozymes. Making copies in the RNA world". Science 292 (5520): 1278. DOI:10.1126/science.292.5520.1278a. PMID 11360970.
-
Szathmáry E (1992). "What is the optimum size for the genetic alphabet?". Proc Natl Acad Sci U S A 89 (7): 2614–8. DOI:10.1073/pnas.89.7.2614. PMID 1372984.
-
Lindahl T (1993). "Instability and decay of the primary structure of DNA". Nature 362 (6422): 709–15. DOI:10.1038/362709a0. PMID 8469282.
-
Vreeland R, Rosenzweig W, Powers D (2000). "Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal". Nature 407 (6806): 897–900. DOI:10.1038/35038060. PMID 11057666.
-
Hebsgaard M, Phillips M, Willerslev E (2005). "Geologically ancient DNA: fact or artefact?". Trends Microbiol 13 (5): 212–20. DOI:10.1016/j.tim.2005.03.010. PMID 15866038.
-
Nickle D, Learn G, Rain M, Mullins J, Mittler J (2002). "Curiously modern DNA for a "250 million-year-old" bacterium". J Mol Evol 54 (1): 134–7. DOI:10.1007/s00239-001-0025-x. PMID 11734907.
-
Birnbaum D, Coulier F, Pébusque MJ, Pontarotti P. (2000). ""Paleogenomics": looking in the past to the future". J Exp Zool. 288 ((1): 21-2). [2]
-
Blanchette M, Green ED, Miller W, Haussler D. (2004). "Reconstructing large regions of an ancestral mammalian genome in silico". Genome Res. 14 ((12): 2412-23). Erratum in: Genome Res. 2005 Mar;15(3):451.[3]
-
Gaucher EA, Thomson JM, Burgan MF, Benner SA. (2003). "Inferring the palaeoenvironment of ancient bacteria on the basis of resurrected protein". Nature 425 ((6955): 285-8). [4]
-
Thornton JW. (2004). "Resurrecting ancient genes: experimental analysis of extinct molecules". Nat Rev Genet. 5 ((5): 366-75). [5]
-
Benner SA, Caraco MD, Thomson JM, Gaucher EA. (2002). "Planetary biology--paleontological, geological, and molecular histories of life". Science 296 ((5569): 864-8). itool=EntrezSystem2.PEntrez.Pubmed.Pubmed_ResultsPanel.Pubmed_RVDocSum
-
Miller WL. (1979). "Use of recombinant DNA technology for the production of polypeptides". Adv Exp Med Biol. 118: 153-74. [6]
-
Leader B, Baca QJ, Golan DE. (2008). "Protein therapeutics: a summary and pharmacological classification". Nat Rev Drug Discov. 7 ((1) 21-39). [7]
-
Dingermann T. (2008). "Recombinant therapeutic proteins: production platforms and challenges". Biotechnol J. 3 ((1) 90-7). [8]
-
Voigt K, Izsvák Z, Ivics Z. (2008). "Targeted gene insertion for molecular medicine". J Mol Med. Jul 8.. [9]
-
Houdebine L (2007). "Transgenic animal models in biomedical research". Methods Mol Biol 360: 163–202. PMID 17172731. [10]
-
Soler E, Thépot D, Rival-Gervier S, Jolivet G, Houdebine LM. (2006). "Preparation of recombinant proteins in milk to improve human and animal health". Reprod Nutr Dev. 46 ((5) 579-88). [11]
-
Chávez A, Muñoz de Chávez M. (2003). "Nutrigenomics in public health nutrition: short-term perspectives". Eur J Clin Nutr. 57 (Suppl 1 S97-100). [12]
-
Vasil IK. (2007). "Molecular genetic improvement of cereals: transgenic wheat (Triticum aestivum L.)". Plant Cell Rep. 26 ((8) 1133-54). [13]
-
Daniell H, Dhingra A (2002). "Multigene engineering: dawn of an exciting new era in biotechnology". Curr Opin Biotechnol 13 (2): 136–41. DOI:10.1016/S0958-1669(02)00297-5. PMID 11950565.
-
Job D (2002). "Plant biotechnology in agriculture". Biochimie 84 (11): 1105–10. DOI:10.1016/S0300-9084(02)00013-5. PMID 12595138.
-
Collins A, Morton N (1994). "Likelihood ratios for DNA identification". Proc Natl Acad Sci USA 91 (13): 6007–11. DOI:10.1073/pnas.91.13.6007. PMID 8016106.
-
Weir B, Triggs C, Starling L, Stowell L, Walsh K, Buckleton J (1997). "Interpreting DNA mixtures". J Forensic Sci 42 (2): 213–22. PMID 9068179.
-
Jeffreys A, Wilson V, Thein S (1985). "Individual-specific 'fingerprints' of human DNA". Nature 316 (6023): 76–9. DOI:10.1038/316076a0. PMID 2989708.
-
Colin Pitchfork — first murder conviction on DNA evidence also clears the prime suspect Forensic Science Service - Acedido a 23 Dez 2006
-
DNA Identification in Mass Fatality Incidents. National Institute of Justice (September 2006).
-
Bhattacharya, Shaoni. "Killer convicted thanks to relative's DNA". newscientist.com (20 de Abril de 2004). Acedido a 22 Dez 06
-
Baldi, Pierre. Brunak, Soren (2001). Bioinformatics: The Machine Learning Approach. MIT Press.
-
Gusfield, Dan. Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology. Cambridge University Press, 15 January 1997. ISBN 978-0-521-58519-4.
-
Sjölander K (2004). "Phylogenomic inference of protein molecular function: advances and challenges". Bioinformatics 20 (2): 170–9. DOI:10.1093/bioinformatics/bth021. PMID 14734307.
-
Mount, DM (2004). Bioinformatics: Sequence and Genome Analysis, 2, Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press.
-
Yin P, Hariadi RF, Sahu S, Choi HMT, Park SH, LaBean T, Reif JH. (2008). "Programming DNA Tube Circumferences". Science 321 ((5890) 824 - 826). [14]
-
Wray G (2002). "Dating branches on the tree of life using DNA". Genome Biol 3 (1): REVIEWS0001. DOI:10.1046/j.1525-142X.1999.99010.x. PMID 11806830.
-
Lost Tribes of Israel, NOVA, PBS airdate: 22 February 2000. Transcript available from PBS.org, (last accessed on 4 March 2006)
-
Kleiman, Yaakov. "The Cohanim/DNA Connection: The fascinating story of how DNA studies confirm an ancient biblical tradition". aish.com (January 13, 2000). Accessed 4 March 2







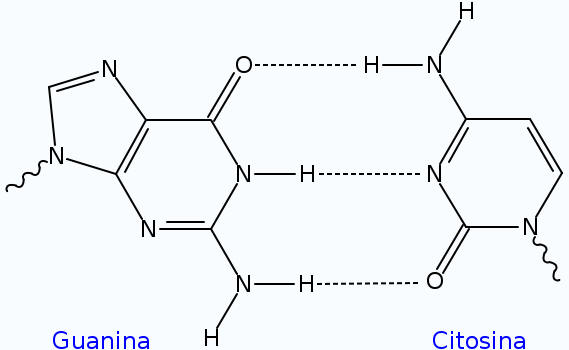



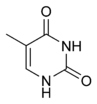

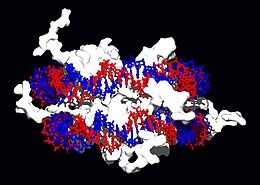
.JPG/260px-Nucleosome_(opposites_attracts).JPG)